

Pre-breeding bei der Kartoffel (Solanum tuberosum) zur Verbesserung der Resistenz gegen Phytophthora infestans
Pre-breeding of potato (Solanum tuberosum) to improve resistance against Phytophthora infestans
Journal für Kulturpflanzen, 72 (12). S. 577–585, 2020, ISSN 1867-0911, DOI: 10.5073/JfK.2020.12.03, Verlag Eugen Ulmer KG, Stuttgart
 | Dies ist ein Open-Access-Artikel, der unter den Bedingungen der Creative Commons Namensnennung 4.0 International Lizenz (CC BY 4.0) zur Verfügung gestellt wird (https://creativecommons.org/licenses/by/4.0/deed.de). This is an Open Access article distributed under the terms of the Creative Commons Attribution 4.0 International License (https://creativecommons.org/licenses/by/4.0/deed.en). |
Die Kraut- und Knollenfäule (Phytophthora infestans Mont. de Bary) verursacht in der Kartoffelproduktion jährlich enorme Ernteverluste. Bislang gibt es dennoch keine zugelassene Sorte, die eine stabile Resistenz mit sich bringt. Am Julius Kühn-Institut konnten in den letzten Jahren neue Zuchtstämme entwickelt werden, die sich bereits in umfangreichen Resistenzuntersuchungen bewährt haben. Damit einhergehend wurden anhand einer genomweiten Assoziationsstudie mit 265 Pre-breeding-Stämmen und zugelassenen Sorten neun Marker für fünf QTL auf den Chromosomen 1, 7, 10 und 11 identifiziert, die mit der Resistenz in Zusammenhang stehen. Diese Ergebnisse können in Zukunft dazu beitragen, die Entwicklung von resistenten Sorten zu vereinfachen.
Stichwörter: Phytophthora infestans, Kraut- und Knollenfäule, Solanum tuberosum, Kartoffel, genomweite Assoziationsstudie
Late blight (Phytophthora infestans Mont. de Bary) causes high yield losses in potato production every year. However, to date there is no approved variety that provides stable resistance. In recent years, pre-breeding clones have been developed at the Julius Kühn Institute that have proven their value in extensive resistance tests. This was accompanied by a genome-wide association study involving 265 pre-breeding clones and approved varieties, respectively, which identified nine markers for five QTL on chromosomes 1, 7, 10, and 11 that are associated with the resistance. These results may help to develop resistant varieties in the future.
Key words: Phytophthora infestans, late blight, Solanum tuberosum, potato, genome-wide association study
Nach wie vor gehört die Kartoffel (Solanum tuberosum L.) zu den wichtigsten Nahrungspflanzen weltweit. Dennoch ist ihre Ertragsstabilität vor allem durch Krankheiten wie die Kraut- und Knollenfäule (Abb. 1–4), hervorgerufen durch den Erreger Phytophthora infestans (P.i.), gefährdet. Bislang mangelt es trotz jahrzehntelanger Forschung an Sorten, welche die zahlreichen für Kartoffeln vermarktungsrelevanten Eigenschaften mit einer hinreichend hohen und dauerhaften Widerstandsfähigkeit gegen P. infestans vereinen würden. Im Jahr 2019 waren in Deutschland sieben Sorten mit der Boniturnote 3 (Noten von 1 (geringe Anfälligkeit) bis 9 (hohe Anfälligkeit)) für den konventionellen Anbau und eine Sorte mit der Note 4 für den ökologischen Anbau registriert (Bundessortenamt, 2019). Die Züchtung auf P.i.-Resistenz wird dadurch erschwert, dass Resistenzen gegen Krautfäule einerseits und Knollenfäule andererseits nach bisherigen Erkenntnissen unabhängig voneinander vererbt werden (Mayton et al., 2010). Im letzten Jahrhundert gab es einige vielversprechende Ansätze für die Nutzung sogenannter R-Gene gegen Krautfäule, die jeweils eine hohe Resistenzausprägung ihrer Träger bedingten. Für diesen qualitativ ausgeprägten Typ der Resistenz sind insgesamt 11 R-Gene, die aus der Wildart S. demissum stammen und in die Kulturkartoffel eingekreuzt wurden, beschrieben worden (Black et al., 1953; Malcolmson & Black, 1966; Malcolmson, 1969). Nachdem inzwischen diese Resistenzen jedoch alle von entsprechenden Pathotypen des sehr anpassungsfähigen Erregers gebrochen worden sind (Collins et al., 1999; Hein et al., 2009), steht heute der quantitativ vererbte Typ der Resistenz im Fokus der Forschung, der durch die Beteiligung mehrerer bis vieler Gene mit jeweils unvollständiger Resistenzwirkung und längere Beständigkeit der Gesamtwirkung gegenüber der Pathogenpopulation gekennzeichnet ist. Am Julius Kühn-Institut (JKI) in Groß Lüsewitz wurden explizit für den ökologischen Anbau, bei dem die Bekämpfung von P.i. aufgrund der geltenden Beschränkung im Einsatz von Kupferpräparaten auf maximal 6 kg Cu2+ je Hektar und Jahr besonders schwierig ist (Verordnung (EG) Nr. 889/2008), im Rahmen eines BÖLN-Projekts (2810 OE 121) von 2012 bis 2018 ältere und jüngere Pre-breeding-Stämme des JKI hinsichtlich ihrer Resistenz- und Qualitätseigenschaften untersucht. Zudem wurde anhand von Sorten und Zuchtstämmen eine genomweite Assoziationsstudie durchgeführt, um QTL und entsprechende Marker für die quantitative P.i.-Resistenz der Zuchtstämme zu identifizieren.

Abb. 1. Gegen Krautfäule anfällige und resistente Stämme im Vergleich vier Wochen nach der Inokulation mit P. infestans.

Abb. 4. Knollenfäulebefall im Scheibentest sechs Tage nach der Inokulation mit P. infestans. Die Scheiben aus Reihe eins und drei stammen von anfälligen Genotypen, dagegen sind in Reihe zwei Scheiben eines resistenten Genotyps gezeigt.
Insgesamt wurden zwischen 2012 und 2018 102 Sorten (Tab. S1), 67 ältere JKI-Pre-breeding-Stämme sowie 166 innerhalb des Projektes am JKI bzw. an der Bayerischen Landesanstalt für Landwirtschaft (LfL) entwickelte Stämme untersucht.
Die Bewertung der P.i.-Resistenz erfolgte durch Erfassung des Befalls nach der Inokulation je Stamm oder Sorte mit vier Methoden: Feldprüfung, Einzelblatttest, Tauchtest und Scheibentest. Alle Methoden wurden je Stamm bzw. Sorte in drei aufeinander folgenden Jahren durchgeführt.
Feldprüfung. Die Erfassung des Krautfäulebefalls erfolgte in einer vollständig randomisierten Blockanlage mit je zwei Wiederholungen à fünf Pflanzen je Stamm bzw. Sorte. Als Windschutz diente ein Mantel aus Hanfpflanzen. Zum Blühende der Sorte ‘Adretta‘ wurde jeweils eine Pflanze je Versuchsparzelle mit 5 ml einer P.i.-Suspension verschiedener Pathotypen inokuliert, deren Zusammensetzung jedes Jahr mit neu gesammeltem Erregermaterial ergänzt wurde (1,2 × 104 Sporangien/ml, Darsow, 2008). Anschließend erfolgte die Bonitur des Befalls in Prozent befallener Krautfläche jeder Parzelle unter Ausschluss der inokulierten Pflanze alle drei bis vier Tage, bis ein Befall von 100 % erreicht war, bzw. bis zur Abreife des jeweiligen Stammes. Anhand dieser Bonituren wurden rAUDPC-Werte (relative Area Under Disease Progress Curve; Fry, 1978; Colon, 1994) berechnet. Da die Krautfäuleresistenz häufig in Zusammenhang mit einer späten Abreife steht, wurden die rAUDPC-Werte anschließend nach Truberg et al. (2009) reifekorrigiert und als ΔrAUDPC-Werte dargestellt.
Einzelblatttest. Kurz vor der Inokulation im Feldversuch wurden jeweils fünf Blätter verschiedener Pflanzen eines Stammes entnommen und im Labor je 20 μl P.i.-Suspension auf die Blattunterseite getropft. Nach 24 h wurden die Blätter gedreht und für weitere fünf Tage bei 16°C, 95 % relativer Feuchte (RF) und 150 Lux inkubiert. Die Bonitur erfolgte anhand der nekrotisierten Blattfläche und der Mycelbildung auf der Blattunterseite (Tab. 1).
Tab. 1. Boniturskala für den Einzelblatttest. Die Bonitur erfolgte sechs Tage nach der Inokulation mit P. infestans.
Note | Luftmyzel | Nekrotisierung |
1 | Kein Myzel | Keine Nekrosen |
2 | Leichte Symptome erkennbar | Punktförmige Nekrosen |
3 | bis zu 4 % der Fläche mit Myzel bedeckt | 2–4 mm große Nekrosen |
4 | 5–12 % der Fläche mit Myzel bedeckt | 5–12 % Zerstörung der Fläche |
5 | 13–30 % der Fläche mit Myzel bedeckt | 13–30 % Zerstörung der Fläche |
6 | 31–55 % der Fläche mit Myzel bedeckt | 31–55 % Zerstörung der Fläche |
7 | 56–78 % der Fläche mit Myzel bedeckt | 56–78 % Zerstörung der Fläche |
8 | 79–96 % der Fläche mit Myzel bedeckt | 79–96 % Zerstörung der Fläche |
9 | 97–100 % der Fläche mit Myzel bedeckt | 97–100 % Zerstörung der Fläche |
Tauchtest. Beim Tauchtest wird die Widerstandsfähigkeit der gesamten Knolle gegenüber Knollenfäule ermittelt. Dafür wurden je Stamm 15 Knollen in eine P.i.-Suspension (2000 Sporangien/ml) getaucht und für 24 h in Dunkelheit bei 19°C/100 % RF inkubiert. Anschließend erfolgte eine 14-tägige Inkubation bei 16°C/85 % RF in Dunkelheit. Der Befall mit Knollenfäule wurde einzelknollenweise erfasst und als Mittelwert je Stamm zusammengefasst (Tab. 2). Knollen, die keinen oder sehr geringen Befall (Note 2) zeigten, wurden nach 14 Tagen erneut bonitiert und gingen mit halber Gewichtung in den Index ein (Darsow, 2008). Diese zweite Bonitur diente dem Auffinden von Knollen, die bei der ersten Bonitur so schwach befallen waren, dass der Befall nicht zu erkennen war. Um diesen späten und im Lager deutlich weniger gefährlichen Befall nicht zu überwerten, erfolgte die Gewichtung im Index mit dem Faktor 0,5.
Tab. 2. Boniturskala für den Tauchtest. Die Bonitur erfolgte 14 Tage nach der Inokulation mit P. infestans.
Note | Verbräunung der Knollenoberfläche |
1 | Keine Verbräunung |
2 | Leichte Verbräunung erkennbar |
3 | bis zu 4 % der Fläche verbräunt |
4 | 5–15 % der Fläche verbräunt |
5 | 16–37 % der Fläche verbräunt |
6 | 38–57 % der Fläche verbräunt |
7 | 58–75 % der Fläche verbräunt |
8 | 76–93 % der Fläche verbräunt |
9 | 94–100 % der Fläche verbräunt |
Scheibentest. Um auch die Resistenzeigenschaften des Knollenmarks zu bewerten, wurde ein Scheibentest zu zwei Zeitpunkten mit vier Knollen je Stamm und Konzentrationsstufe durchgeführt. Von jeder Knolle wurde jeweils eine Scheibe an beiden Terminen mit je 20 μL einer niedrig (1.900 Sporangien/ml) bzw. einer hoch (15.000 Sporangien/ml) konzentrierten P.i.-Suspension inokuliert. Nach 24 h wurden die Scheiben gedreht und anschließend für weitere fünf Tage bei 16°C/95 % RF inkubiert. Für die Bonitur wurden die Scheiben mit jener P.i.-Konzentration gewertet, die eine Differenzierung des Befalls ermöglichte (Tab. 3).
Tab. 3. Boniturskala für den Scheibentest. Die Bonitur erfolgte sechs Tage nach der Inokulation mit P. infestans.
Note | Luftmyzel | Verbräunung |
1 | Kein Myzel | Keine Verbräunung |
2 | Leichte Symptome erkennbar | Leichte Verbräunung erkennbar |
3 | bis zu 5 % der Fläche mit Myzel bedeckt | bis zu 14 % der Fläche verbräunt |
4 | 6–12 % (Mittel 9 %) der Fläche mit Myzel bedeckt | 15–24 % der Fläche verbräunt |
5 | 13–40 % (Mittel 25 %) der Fläche mit Myzel bedeckt | 25–44 % der Fläche verbräunt |
6 | 31–60 % (Mittel 43 %) der Fläche mit Myzel bedeckt | 45–66 % der Fläche verbräunt |
7 | 50–85 % (Mittel 62 %) der Fläche mit Myzel bedeckt | 67–82 % der Fläche verbräunt |
8 | 75–96 % (Mittel 85 %) der Fläche mit Myzel bedeckt | 83–96 % der Fläche verbräunt |
9 | 93–100 % (Mittel 96 %) der Fläche mit Myzel bedeckt | 97–100 % der Fläche verbräunt |
Die Auswertungen der Laboruntersuchungen wurden neben den ΔrAUDPC-Werten für die Selektion von geeigneten Zuchtstämmen verwendet. Es wurde zudem geprüft, ob die Ergebnisse der verschiedenen Tests nach Pearson miteinander korrelieren. Prüfglieder, die sowohl in der Feldprüfung als auch in den Labortests eine hinreichende P.i.-Resistenz zeigten (durchschnittliche Boniturnoten < 5, ΔrAUDPC-Werte < 0), wurden im dritten Versuchsjahr auch hinsichtlich ihrer Speiseeignung untersucht (Daten unveröffentlicht).
Die Auswertung der Labor- und Felddaten durch eine Varianzanalyse mit einem gemischten linearen Modell erfolgte mit dem Programm „R“. Hierbei wurden die Jahre als Umwelten betrachtet:
yijk |
= |
μ + Gi + Ej + GxEij + ɛijk |
|
|
|
yijk |
= |
ΔrAUDPC-Wert bzw. Note des Scheiben- Blatt- oder Tauchtests des i-ten Stamms in der j-ten Umwelt und der k-ten Wiederholung |
Gi |
= |
i-ter Stamm |
Ej |
= |
Umwelt j für drei verschiedene Jahre |
GxEij |
= |
Interaktion des Stammes und der Umwelt |
ɛijk |
= |
Restfehler |
Die Heritabilität des Merkmals ΔrAUDPC wurde nach Holland et al. (2003) berechnet.
Um molekulare Marker für die quantitative P.i.-Resistenz der JKI-Zuchtstämme zu identifizieren, wurde von 265 ausgewählten Stämmen und Sorten Blattmaterial entnommen und nach der CTAB-Methode (Stein et al., 2001) DNA extrahiert.
Anschließend erfolgte die Genotypisierung mit der DArTSeq Plattform (Erhalt von DArT und SNP Markern) durch Diversity Arrays Technology Pty Ltd, Canberra, Australien (https://www.diversityarrays.com/). Marker mit einer Minor-Allelfrequenz < 5 % oder einem Anteil an doppelten Nullallelen > 20 % wurden nicht berücksichtigt. Fehlende Markerdaten wurden mit der K-Nearest Neighbours-Methode ersetzt (Batista & Monard, 2002).
Für die Assoziationsstudie wurden LS-Means (Least Square Means) der ΔrAUDPC-Werte berechnet und mit den Daten der Genotypisierung als Alleldosis in R mit dem Modell „Diplo-additive“ (Paket „GWASpoly“ nach Rosyara et al., 2016) verrechnet.
Über jeweils drei Jahre wurden Sorten sowie ältere und neuere Pre-breeding-Stämme erfolgreich in ihrer P.i.-Resistenz charakterisiert.
In Abbildung 5 sind Boxplots von 103 Prüfgliedern der Jahre 2015–2017 in ihren ΔrAUDPC-Werten und ihren Reifegruppen im Vergleich gezeigt. Negative ΔrAUDPC-Werte (unterhalb der roten Linie) weisen einen besonders niedrigen reifekorrigierten Krautfäulebefall auf. Der Vergleich zeigt, dass der überwiegende Teil der JKI Stämme deutlich geringer befallen wurde als die meisten zugelassenen Sorten und Zuchtstämme anderer Herkunft und zudem ein großer Teil der relativ resistenten JKI-Stämme der frühen bis mittelfrühen Reifegruppe angehörte. Von den Sorten zeigten mit wenigen Ausnahmen diejenigen einen geringen Befall, die spät abreiften (Abb. 5).
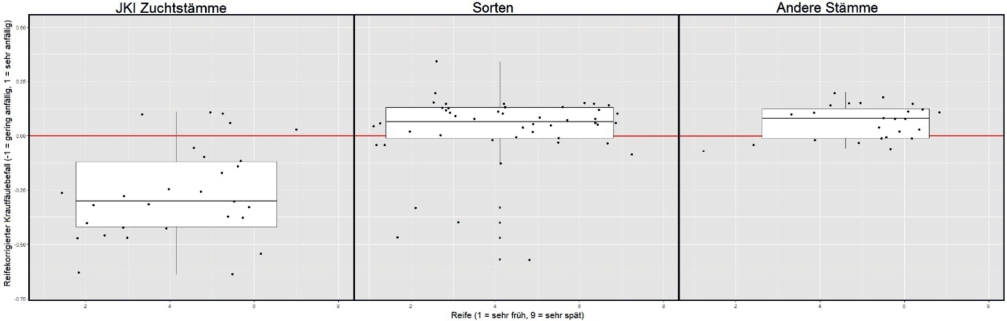
Abb. 5. ΔrAUDPC-Werte und Reifeboniturnoten von Sorten und Zuchtstämmen im Mittel von drei Jahren. Die Boxplots zeigen jeweils den Median (dicke waagerechte Linie), das obere und untere Quartil (dünne waagerechte Linien) sowie Minimum und Maximum (Ende der Whisker) unter Ausschluss der Ausreißer. In Rot ist die Nulllinie dargestellt.
Auch die Ergebnisse der Labortests wiesen große Unterschiede zwischen den Sorten und Zuchtstämmen auf. Bei sechs Stämmen war eine besonders geringe Ausbreitung der Kraut- und Knollenfäule zu beobachten. Zugleich konnten diese Stämme frühen bis mittelfrühen Reifegruppen zugeordnet werden. Die Sorten wiesen auch bei den Labortests eine erhöhte Anfälligkeit auf (Tab. 4). Eine Ausnahme stellte die Sorte ‘Sarpo Mira‘ dar, die zwar gute Resistenzeigenschaften mit sich bringt, aber für den deutschen Markt deutlich zu spät abreift.
Tab. 4. Pi.-Befall an drei älteren und drei neu entwickelten Zuchtstämmen sowie vier Sorten im dreijährigen Mittel. Boniturskalen a) 1 = kein Befall, 9 = Blattfläche vollständig nekrotisiert und mit Myzel bedeckt, b) 1 = kein Befall, 9 = 100 % Zerfall des Knollengewebes, c) 1 = kein Befall, 9 = Verbräunung der Scheiben, mit Mycel bedeckt, d) 1 = sehr früh, 9 = sehr spät
Stamm bzw. Sorte | rAUDPC [%] | ΔrAUDPC | Blatttest [Notea)] | Knollentauchtest[Indexb)] | Scheibentest | Reife [Noted)] |
12797/9 | 0 | –0,38 | 2,2 | 1,00 | 2,7 | 5,5 |
12.1022/3 | 2 | –0,38 | 1,2 | 2,10 | 2,4 | 4,1 |
12.1060/2 | 3 | –0,41 | 1,1 | 2,30 | 3,1 | 3,7 |
GL-03.5129.06 | 3 | –0,24 | 1,4 | 1,00 | 2,1 | 5,3 |
GL-04.5170.02 | 0 | –0,26 | 1,5 | 3,30 | 4,0 | 6,3 |
GL-04.5230.06 | 7 | –0,23 | 1,3 | 1,55 | 3,0 | 3,9 |
Adretta | 63 | 0,32 | 4,5 | 5,61 | 5,8 | 3,6 |
Jelly | 40 | 0,14 | 3,4 | 5,10 | 7,3 | 6,3 |
Princess | 54 | 0,26 | 6,0 | 4,10 | 7,6 | 5,6 |
Sarpo Mira | 2 | –0,23 | 2,3 | 4,15 | 6,5 | 6,8 |
Grenzdifferenz 5 % | 15 | 0,08 | 1,0 | 0,2 | 0,3 |
|
Die Ergebnisse der verschiedenen Labortests und der Feldprüfung wiesen eine hohe Korrelation auf (Tab. 5). Einzig die Reife war mit keinem weiteren Merkmal korreliert.
Tab. 5. Korrelation der mittleren Feld- und Laborergebnisse nach Pearson.
| Reife | rAUDPC | ΔrAUDPC | Blatttest | Tauchtest |
rAUDPC | –0,70 |
|
|
|
|
ΔrAUDPC | 0,00 | 0,90*** |
|
|
|
Blatttest | –0,03 | 0,65*** | 0,71*** |
|
|
Tauchtest | 0,07 | 0,61*** | 0,57*** | 0,53*** |
|
Scheibentest | –0,05 | 0,55*** | 0,60*** | 0,66*** | 0,58*** |
*** p < 0,001 | |||||
Bei der Testung auf Speiseeignung schnitt der Stamm 12797/9 vergleichsweise gut ab. Die übrigen Stämme waren insgesamt für Speisezwecke wenig geeignet.
Die Varianzanalyse ergab, dass die Stämme in ihren Laborwerten und ΔrAUDPC-Werten signifikante Unterschiede aufwiesen. Die Heritabilität der für die Assoziationsstudie genutzten ΔrAUDPC-Werte lag bei h2 = 0,80.
Für die Assoziationsstudien wurden nach einer Filterung insgesamt 27.710 polymorphe Marker genutzt. In Abbildung 6 ist die Verteilung der Marker über die 12 Chromosomen der Kulturkartoffel dargestellt. Neun Marker mit geringen Markereffekten zwischen –0,13 und 0,12 waren mit dem Merkmal ΔrAUDPC-Wert assoziiert (-log10(p)> 3,5). Sie befinden sich auf den Chromosomen 1, 7, 10 und 11 (Abb. 6).
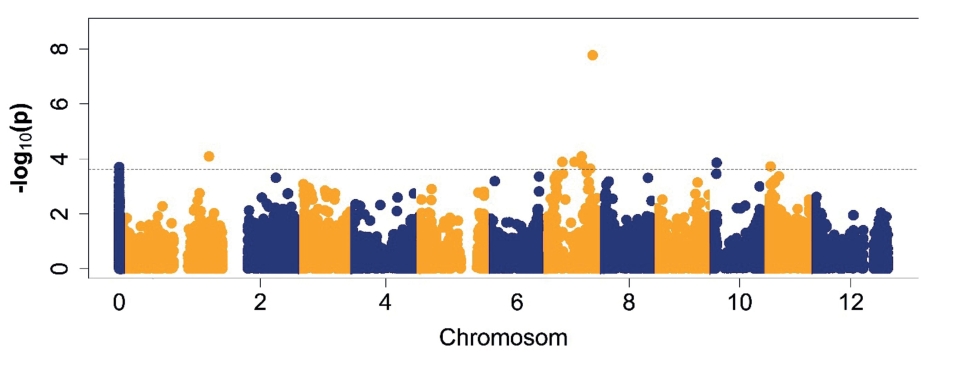
Abb. 6. Manhattan-Plot (diplo-additive) für das Merkmal ΔrAUDPC. Das Signifikanzniveau ((-log10(p)> 3,5) wurde durch die Kontrolle der False Discovery Rate von 5 % festgelegt. Die Differenzierung der Chromosomen ist durch die abwechselnde Färbung gegeben.
Die Ergebnisse zeigen, dass Stämme entwickelt werden konnten, die sich in ihrer P.i.-Resistenz deutlich von den derzeit zugelassenen Sorten unterscheiden. Ein Stamm (12797/9) zeigte gute Ergebnisse auch bei der Speisewertprüfung. Dies verdeutlicht, dass das Einkreuzen von Sorten zur Verbesserung der agronomischen Merkmale der Pre-breeding-Stämme in den nächsten Jahren eine wichtige Rolle spielt, um diese in einer anschließenden Sortenzüchtung verwenden zu können.
Da die Ergebnisse der Feld- und Labortests stark miteinander korrelieren, wäre es denkbar den Testumfang für zukünftige Stämme zu reduzieren. Eine Möglichkeit wäre, statt je zwei Tests für Kraut- und Knollenfäule nur noch jeweils einen Test zu verwenden.
Die genomweite Assoziationsstudie zeigt fünf QTL mit kleinen Effekten für das Merkmal P.i.-Resistenz der untersuchten Zuchtstämme auf. Diese niedrige Zahl an Resistenzfaktoren wirft die Frage auf, ob es sich bei den QTL um R-Gene handeln könnte. Gegen eine solche Annahme spricht die Pathotypen-Zusammensetzung des in dieser Studie verwendeten Inokulums, welches alle elf bekannten R-Gene aus S. demissum überwindet. Die in dieser Studie beobachteten, resistenzwirksamen QTL repräsentieren somit wahrscheinlich anderweitige Resistenzfaktoren. Die durch diese Faktoren bedingte Krautfäuleresistenz der betroffenen Zuchtstämme ist in unseren Feldprüfungen bislang über mindestens vier Jahre wirksam geblieben und dürfte somit einen dauerhaften Resistenztypus darstellen.
Danan et al. (2011) schließen aus ihrer Metaanalyse von 19 unabhängigen Studien zur QTL-Kartierung von P.i.-Resistenz, dass resistenzbedingte QTL auf allen zwölf Chromosomen des Kartoffelgenoms vorliegen. In den einzelnen Analysen wurden jeweils, wie auch in dieser Studie, nur vereinzelte QTL nachgewiesen. Daraus lässt sich ableiten, dass die gesamte genetische Diversität der Kartoffel und ihrer kreuzbaren Wildarten sehr viele verschiedenen Resistenzquellen bereithält und es weiterer Forschung bedarf, die Wirksamkeit und Dauerhaftigkeit der betreffenden QTL zu untersuchen und züchterisch zu erschließen.
Mithilfe von Markern, die mit dem Merkmal ΔrAUDPC assoziiert sind, wäre es denkbar, bereits Pflanzen im Sämlingsstadium (F1) auf P.i-Resistenz zu selektieren. Da sich die Assoziation in dieser Studie allerdings als schwach erwies, sind weitere Studien angedacht.
Die neuen Stämme bieten sich besonders für den ökologischen Landbau an, da die Bekämpfung von P.i. hier nur schwer möglich ist. Aber auch der konventionelle Kartoffelanbau könnte von neuen Sorten profitieren, deren Resistenzniveau durch Einkreuzung der hier beschriebenen Pre-breeding-Stämme verbessert ist. Die hierdurch mögliche Reduzierung der chemischen Pflanzenschutzmaßnahmen wäre in künftigen Versuchen zu thematisieren. Das Pre-breeding-Material kann zudem der Erweiterung des Genpools der europäischen Kulturkartoffel dienen, weil es bislang in der Sortenzüchtung nicht genutzte genetische Diversität aus Solanum-Wildarten enthält, die auf konventionellem züchterischem Weg eingekreuzt worden ist. Die zur Verfügung stehenden Zuchtstämme bieten eine Grundlage, in Zukunft Sorten mit verbesserter Resistenz gegen die Kraut- und Knollenfäule bei nicht zu später Abreife und guter Speiseeignung zu entwickeln.
Die in den bisherigen und folgenden Arbeiten generierten Daten zu molekularen Markern sollen in weiteren Analysen auch auf potentielle Zusammenhänge zur Knollenfäule untersucht werden. Überdies werden die beschriebenen sowie weitere Zuchtstämme und Neuzüchtungen neben der P.i.-Resistenz auch auf ihre Nährstoffeffizienz untersucht (Projekt EffiKar).
Die Autoren danken der Bayerischen Landesanstalt für Landwirtschaft und den beteiligten Kartoffelzüchtern für die Bereitstellung von Stämmen und Sorten. Das Vorhaben wurde aus Mitteln des Bundesministeriums für Ernährung und Landwirtschaft (BMEL) im Bundesprogramm Ökologischer Landbau und andere Formen nachhaltiger Landwirtschaft (BÖLN 2810 OE 121) unter der Projektträgerschaft der Bundesanstalt für Landwirtschaft und Ernährung (BLE) finanziert.
Die Autoren erklären, dass keine Interessenskonflikte vorliegen.
Batista, G.E.A.P.A., M.C. Monard, 2002: A Study of K-Nearest Neighbour as an Imputation Method. HIS 87 (48), 1-10.
Black, W., C. Mastenbroek, W.R. Mills, L.C. Peterson, 1953: A proposal for an international nomenclature of races of Phytophthora infestans and of genes controlling immunity in Solanum demissum derivatives. Euphytica 2 (3), 173–179, DOI: 10.1007/BF00053724.
Bundessortenamt (Hrsg.), 2019: Beschreibende Sortenliste Kartoffel, 2019, Hannover, ISSN: 14 30-97 77.
Collins, A., D. Milbourne, L. Ramsay, R. Meyer, C. Chatot-Balandras, P. Oberhagemann, W. De Jong, C. Gebhard, E. Bonnel, R. Waugh, 1999: QTL for field resistance to late blight in potato are strongly correlated with maturity and vigour. Molecular Breeding 5 (5), 387-398.
Colon, L., 1994: Resistance to Phytophthora infestans in Solanum tuberosum and wild Solanum species. Dissertation Agricultural University Wageningen, 1-159.
Danan, S., J.B. Veyrieras, V. Lefebvre, 2011: Construction of a potato consensus map and QTL meta-analysis offer new insights into the genetic architecture of late blight resistance and plant maturity traits. BMC Plant Biology 11 (1), 1–16, DOI: 10.1186/1471-2229-11-16.
Darsow, U., 2008: Vorlaufzüchtung der Kartoffel auf quantitative Phytophthora-Resistenz im ILK Groß Lüsewitz in der Ressortforschung des BMELV. Stand der Forschung und Züchtung. Mitteilungen aus dem Julius Kühn-Institut 415, 1-128.
Fry, W.E., 1978: Quantification of general resistance of potato cultivars and fungicide effects for integrated control of potato late blight. Phytopathology 68 (11), 1650-1655.
Hein, I., P.R. Birch, S. Danan, V. Lefebvre, D.A. Odeny, C. Gebhardt, F. Trognitz, G.J. Bryan, 2009: Progress in mapping and cloning qualitative and quantitative resistance against Phytophthora infestans in potato and its wild relatives. Potato Research 52 (3), 215–227, DOI: 10.1007/s11540-009-9129-2.
Holland, J.B., W.E. Nyquist, C.T. Cervantes-Martínez, 2003: Estimating and interpreting heritability for plant breeding: an update. Plant Breeding Reviews 22, 1-104.
Malcolmson, J.F., 1969: Races of Phytophthora infestans occurring in Great Britain. Transactions of the British Mycological Society 53 (3), 417–423, DOI: 10.1016/S0007-1536(69)80099-9.
Malcolmson, J.F., W. Black, 1966: New R genes in Solanum demissum Lindl. and their complementary races of Phytophthora infestans (Mont.) de Bary. Euphytica 15 (2), 199–203, DOI: 10.1007/BF00022324.
Mayton, H., H. Griffiths, I. Simko, S. Cheng, J. Lorenzen, W. De Jong, W.E. Fry, 2010: Foliar and tuber late blight resistance in a Solanum tuberosum breeding population. Plant Breeding 129 (2), 197–201, DOI: 10.1111/j.1439-0523.2009.01671.x.
Rosyara, U.R., W.S. De Jong, D.S. Douches, J.B. Endelman, 2016: Software for genome-wide association studies in autopolyploids and its application to potato. The Plant Genome 9 (2), DOI: 10.3835/plantgenome2015.08.0073.
Stein, N., G. Herren, B. Keller, 2001: A new DNA extraction method for high-throughput marker analysis in a large-genome species such as Triticum aestivum. Plant Breeding 120 (4), 354-356.
Truberg, B., T. Hammann, U. Darsow, H.P. Piepho, 2009: Empirischer Vergleich verschiedener Methoden zur Reifekorrektur von Daten zum Befall mit Krautfäule (Phytophthora infestans (Mont.) de Bary) in Selektionsexperimenten bei der Kartoffel (Solanum tuberosum subsp. tuberosum). Journal für Kulturpflanzen – Journal of Cultivated Plants 61 (3), 77-81.
Tab. S1. Zwischen 2012 und 2018 untersuchte Kartoffelsorten.
lfd-Nr. | Sorte | lfd-Nr. | Sorte | lfd-Nr. | Sorte |
1 | Adretta | 35 | Eurobeta | 69 | Novago |
2 | Agila | 36 | Fidelia | 70 | Omega |
3 | Agria | 37 | Figaro | 71 | Opal |
4 | Allians | 38 | Flamingskost | 72 | Osira |
5 | Allora | 39 | Francisca | 73 | Preciosa |
6 | Alouette | 40 | Goldmarie | 74 | Princess |
7 | Ampera | 41 | Golf | 75 | Quadriga |
8 | Andrea | 42 | Grandifolia | 76 | Queen Anne |
9 | Annalena | 43 | Hansa | 77 | Ramona |
10 | Anuschka | 44 | Heidi | 78 | Red Fantasy |
11 | Axona | 45 | Husar | 79 | Regina |
12 | Baltic Cream | 46 | Jelly | 80 | Ribera |
13 | Barbara | 47 | Juligelb | 81 | Rita |
14 | Belana | 48 | Karlena | 82 | Salome |
15 | Belinda | 49 | Kifli | 83 | Salute |
16 | Bintje | 50 | Kolibri | 84 | Samson |
17 | Biogold | 51 | Königsmark | 85 | Sarpo Mira |
18 | Bionica | 52 | Kuras | 86 | Sirius |
19 | Birgit | 53 | Lady Claire | 87 | Solist |
20 | Blaue Sankt Galler | 54 | Laura | 88 | Solo |
21 | Blue Danube | 55 | Laurette | 89 | Sonja |
22 | Caprice | 56 | Linzer Speise | 90 | Soraya |
23 | Cardinia | 57 | Lisa | 91 | Stefanie |
24 | Carolus | 58 | Lolita | 92 | Steffi |
25 | Cascada | 59 | Margit | 93 | Tacoma |
26 | Colomba | 60 | Mariola | 94 | Talent |
27 | Connect | 61 | Mariska | 95 | Tessa |
28 | Cosma | 62 | Markies | 96 | Toluca |
29 | Diplomat | 63 | Marli | 97 | Tomensa |
30 | Ditta | 64 | Merlot | 98 | Torenia |
31 | Eersteling | 65 | Montana | 99 | Troja |
32 | Elfe | 66 | Musica | 100 | Twinner |
33 | Escort | 67 | Nicola | 101 | Vitabella |
34 | Esprit | 68 | Nordlicht | 102 | Wega |