

Die Bedeutung von RNA-Interferenz (RNAi) als neues Wirkprinzip für den Pflanzenschutz mit hoher Spezifität
Relevance of RNA interference (RNAi) as a new mode of action for plant protection with high specificity
Journal für Kulturpflanzen, 73 (1-2). S. 1–8, 2021, ISSN 1867-0911, DOI: 10.5073/JfK.2021.01-02.02, Verlag Eugen Ulmer KG, Stuttgart
 | Dies ist ein Open-Access-Artikel, der unter den Bedingungen der Creative Commons Namensnennung 4.0 International Lizenz (CC BY 4.0) zur Verfügung gestellt wird (https://creativecommons.org/licenses/by/4.0/deed.de). This is an Open Access article distributed under the terms of the Creative Commons Attribution 4.0 International License (https://creativecommons.org/licenses/by/4.0/deed.en). |
Aufgrund von verschiedenen Herausforderungen und Restriktionen sind im Bereich Pflanzenschutz neue Lösungsansätze gefordert, um zukünftigen Schädlingsproblemen zu begegnen. Eine mögliche Alternative zu konventionellen chemischen Pflanzenschutzmitteln sind Produkte, die auf dem natürlicherweise in Tieren und Pflanzen vorhandenen Kontroll- und Abwehrmechanismus der RNA-Interferenz (RNAi) beruhen. Praktische Verwendung fand RNAi bisher unter anderem bei der Erzeugung gentechnisch veränderter Pflanzen mit Virusresistenz und mit eingebautem Schutz gegen Insekten. In den letzten Jahren hat aber auch der Anwendungsbereich von extern applizierten RNA-basierten Pflanzenschutzmitteln zunehmend an Bedeutung gewonnen, und einige derartige Produkte sind schon weit in der Entwicklung. Um den sicheren und nachhaltigen Einsatz dieses neuen, durch hohe Spezifität gekennzeichneten Wirkprinzips im Pflanzenschutz zu ermöglichen, sind geeignete Regulierungsstandards und angepasste Testmethoden zur Risikoanalyse notwendig.
Stichwörter: RNA-Interferenz, Pflanzenschutz, dsRNA, Biotechnologie
Different challenges and restrictions require new solutions for plant protection in order to meet future pest and pathogen problems. Products based on the mechanism of RNA interference (RNAi), which is naturally present in animals and plants, offer a possible alternative to conventional chemical pesticides. Up to now, RNAi has been the basis of genetically modified plants with virus resistance and with plant incorporated protection against insects. In recent years the range of applications has been extended to externally applied RNA-based products, some of which are already quite advanced in development. In order to enable the safe and sustainable use of this new highly specific mode of action for plant protection, appropriate regulations and adapted test methods for risk analysis are required.
Key words: RNA interference, plant protection, dsRNA, biotechnology
Angesichts des erhöhten Nahrungsmittelbedarfs der wachsenden Weltbevölkerung sowie neuer Herausforderungen aufgrund des Klimawandels, wie neu auftretender Schädlinge und Pflanzenkrankheiten (Garrett et al., 2006; Chakraborty & Newton, 2011), gibt es weltweit einen hohen Bedarf an innovativen Lösungen für den Pflanzenschutz. Bisherige, weitgehend auf chemischen Wirkstoffen basierende, Kontrollmaßnahmen sind nur begrenzt zukunftsfähig, da immer weniger Pestizide zur Verfügung stehen – zum einen wegen negativer Auswirkungen von Insektiziden auf Nützlinge wie etwa Bienen (Wernecke et al., 2019), zum anderen dadurch, dass Schadorganismen Resistenzen gegen Pestizide entwickelt haben (Alyokhin et al., 2008; Rupp et al., 2017). Eine Alternative zu chemischen Pflanzenschutzmitteln ist die Verwendung entsprechend resistenter Sorten, deren Erzeugung mittels konventioneller Pflanzenzüchtung allerdings nur in begrenztem Umfang realisierbar ist. Daher sind neue Ansätze notwendig, um zukünftige Schädlingsprobleme zu bekämpfen.
Im Bereich der Pflanzenzüchtung bieten gentechnische Verfahren eine Möglichkeit, Pflanzen gezielt widerstandsfähig gegen Schädlinge zu machen. Gentechnisch veränderter (GV) Mais, in dessen Erbmaterial das Gen für ein spezifisches Insektentoxin aus dem Bodenbakterium Bacillus thuringiensis eingebaut wurde (Bt-Mais), ist dafür ein Beispiel. Dieser GV-Mais wird in vielen Ländern angebaut, unter anderem in Spanien und Portugal (ca. 120.000 ha). Allerdings fehlt in Europa insgesamt die Akzeptanz für GV-Pflanzen, so dass diese Züchtungstechnologie derzeit keine realistische Option für Europa ist. Auch die zielgerichtete Mutagenese ist als neue Pflanzenzüchtungstechnik in Europa zurzeit nur schwer realisierbar, da zugrundeliegende Methoden (z. B. CRISPR/Cas-Technologie) entsprechend dem Urteil des Europäischen Gerichtshofs vom 25. Juli 2018 (Rechtssache C-528/16) unter die strengen Auflagen der Gentechnik-Regulierung fallen und dementsprechend aufwändige Zulassungsverfahren durchlaufen müssen.
Seit einigen Jahren gibt es zahlreiche Forschungsaktivitäten hinsichtlich der Entwicklung von Ribonukleinsäure (RNA)-basierten Pflanzenschutzmitteln als Alternative zu herkömmlichen chemischen Wirkstoffen (Taning et al., 2019). Das erste Zulassungsverfahren für ein auf dem Wirkmechanismus RNA-Interferenz (RNAi) beruhendes Produkt zur Kontrolle von Kartoffelkäfern läuft bereits in den USA, so dass diese Technologie möglicherweise in absehbarer Zeit auch in Deutschland für praktische Pflanzenschutz-Anwendungen zur Verfügung stehen könnte. Vorteile gegenüber konventionellen Pflanzenschutzmitteln liegen insbesondere in der hohen, auf RNA-Sequenzhomologien beruhenden Spezifität (Bachmann et al., 2013) sowie in der guten Abbaubarkeit von RNA in der Umwelt (Bachmann et al., 2020). Nachfolgend wird der RNAi zugrundeliegende Mechanismus erklärt, mögliche Anwendungsbereiche vorgestellt sowie Fragen der Sicherheitsbewertung und Regulation betrachtet.
Ein alternatives Wirkprinzip, das schon seit längerem in verschiedenen GV-Pflanzen zum Einsatz kommt, ist die RNA-Interferenz (RNAi). Diese beruht auf einem natürlicherweise in Pflanzen und Tieren vorhandenen „Silencing“-Mechanismus, dessen Entdeckung (Fire et al., 1998) 2006 mit einem Nobelpreis für Physiologie oder Medizin honoriert wurde, und der zur Genregulation unter anderem im Lauf der Pflanzenentwicklung sowie zur Abwehr von Viren dient.
RNA-Interferenz basiert ursächlich auf der sequenzspezifischen Interaktion von kleinen RNAs, zu denen micro RNAs (miRNAs) und kleine interferierende RNAs (siRNAs) gehören, mit komplementärer Ziel-RNA (Fire et al., 1998; Baulcombe, 2004; Vaucheret, 2006). Initiiert wird RNAi typischerweise ausgehend von doppelsträngiger RNA (dsRNA), die durch spezifische TypIII-RNasen (sogenannte Dicer- oder DCL-Enzyme) zu siRNAs prozessiert wird. dsRNA wird in Tieren und Pflanzen unter bestimmten Bedingungen ausgehend vom eukaryotischen Genom gebildet, unter anderem durch Transkription von miRNA-Genen (Axtell et al., 2011), entsteht aber auch als Zwischenprodukt während der Replikation des Genoms von einzelsträngigen RNA-Viren (Hamilton & Baulcombe, 1999) sowie ausgehend von komplementären sense- und antisense-Transkripten von DNA-Viren (Blevins et al., 2006). Von den durch Prozessierung entstandenen kleinen RNAs wird ein Strang in einen RNA-induzierten Silencing-Komplex (RISC) eingebaut, was in einem sequenzspezifischen Abbau von komplementärer Ziel-RNA und damit einer Reduktion der Ausprägung der entsprechenden genetischen Information resultiert. Ziel-Molekül der 21 bis 22 Basenpaare (bp) „großen“ siRNAs kann sowohl transkribierte Boten-RNA (mRNA) als auch Virus-RNA sein. Im Fall von Pflanzenviren wird durch RNAi das Virus-Genom degradiert; allerdings besitzen Viren zum Ausschalten der Wirtsabwehr verschiedene Suppressor-Mechanismen, so dass in der Natur ein Wettlauf zwischen erfolgreicher Virusinfektion und RNAi-vermittelter Virusresistenz stattfindet. Abbildung 1 gibt einen Überblick über die Entstehung und die Funktionen von miRNA sowie einer Auswahl von siRNAs in Pflanzen.
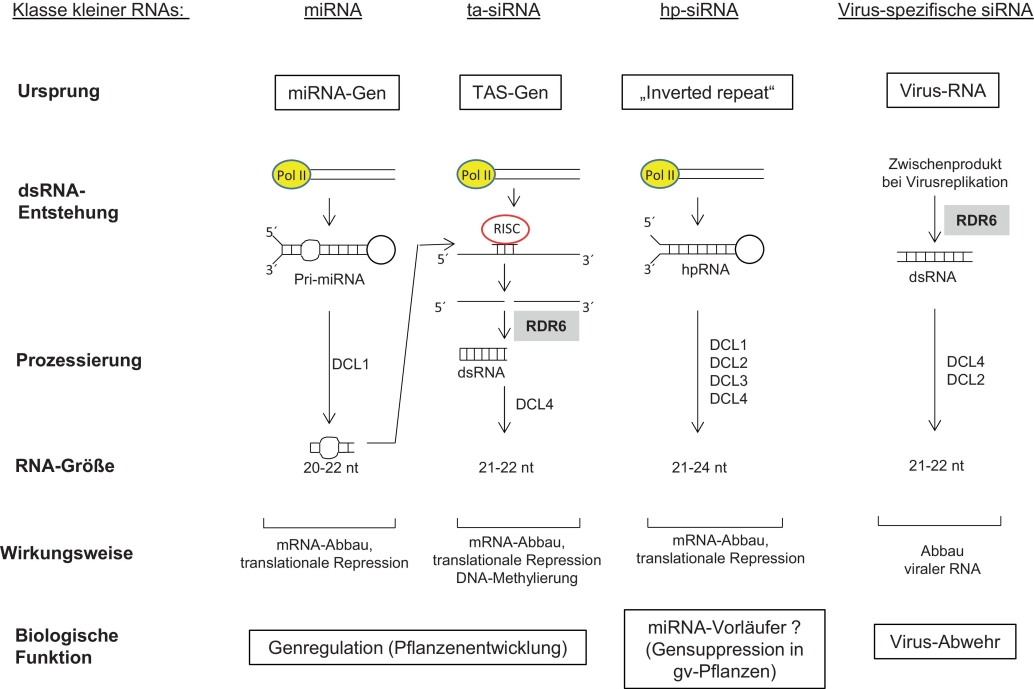
Abb. 1. Überblick über verschiedene RNAi-Mechanismen in Pflanzen. Micro RNAs (miRNAs) entstehen nach sukzessiver Prozessierung durch DCL1 aus pflanzeneigenen Transkripten mit imperfekter doppelsträngiger „stem-loop“-Struktur, sogenannter pri-miRNA (primärer miRNA). Pflanzliche miRNAs zielen auf Boten-RNA mit fast perfekter Sequenzhomologie und führen zum Abbau von Boten-RNA oder zur Hemmung der Translation. MiRNAs entfalten ihre Wirkung zum Teil über sekundäre trans-agierende siRNAs (ta-siRNAs), indem sie ein TAS-Gentranskript ansteuern, spalten und die Spaltungsprodukte als Matrize für die Synthese weiterer dsRNA verwenden. „Hairpin“ (hp) siRNA stammt von langen hpRNAs, die vermutlich Vorläufer von miRNAs sind; hp-siRNA wird auch in entsprechenden gentechnisch veränderten RNAi-Pflanzen gebildet. Während der Virusreplikation entstehende dsRNA wird in siRNAs prozessiert, die eine spezifische antivirale Abwehr gegen infizierende Viren mit homologer RNA auslösen. Pol II = RNA polymerase II, RDR6 = RNA-abhängige RNA-Polymerase 6, DCL = Dicer like RNasen. Abbildung basiert auf der Übersicht der Gen-Silencing-Wege in Schiemann et al. (2019 ).
Bei Kulturpflanzen gibt es mehrere Beispiele von spontan auftretenden RNAi-vermittelten Merkmalen, die während des konventionellen Züchtungsprozesses selektioniert wurden, wie beispielsweise Sojabohnen mit veränderter Farbe der Samenschale (Tuteja et al., 2004) oder Reis mit niedrigem Glutelin-Gehalt (Kusaba et al., 2003). Neben diesen natürlich vorkommenden RNAi-Pflanzenvarianten wurden auch auf gentechnischem Weg zahlreiche Pflanzenlinien erzeugt, in denen aufgrund der gentechnischen Veränderung dsRNA gebildet wird. Prominente Beispiele sind die bereits in den 1990er Jahren vermarktete „Anti-Matsch“- FLAVR SAVR-Tomate (Redenbaugh et al., 1993) sowie die in neuerer Zeit entwickelten und in USA und Kanada für den Anbau zugelassenen Kartoffeln und Äpfel, die aufgrund der RNAi-vermittelten Suppression der Polyphenol-Oxidase-Gene keine unerwünschten braunen Flecken entwickeln (Waltz, 2015).
Im Bereich Pflanzenschutz wurde das RNAi-Wirkprinzip bisher insbesondere zur Herstellung virusresistenter Pflanzen verwendet (Ibrahim & Aragão, 2015). Durch die Bildung von virusspezifischer dsRNA bereits vor Infektion der Pflanze wird dabei der oben erwähnte Wettlauf zwischen Virus und Pflanze zugunsten der Pflanze entschieden. Beispiele sind die seit 1997 in USA zugelassenen und insbesondere in Hawaii angebauten Papaya-Sorten „Rainbow“ und „SunUp“, die gegen das Papaya-Ringfleckenvirus (PRSV) resistent sind (Gonsalves et al., 2006). Da kleine RNAs auch zwischen Pathogenen oder Parasiten und ihrem Wirt transportiert werden können (Wang et al., 2017), ist es zudem möglich, durch gentechnische Veränderung in Pflanzen dsRNA zu produzieren, die gegen essentielle Gene eines extrazellulären Pathogens oder Parasiten gerichtet ist (host-induced gene silencing; HIGS). Auch Insektenschädlinge können durch Wirt-induzierte Gen-Suppression (HIGS) spezifisch bekämpft werden, indem etwa die Larven beim Fressen an der Pflanze dsRNA aufnehmen, die dann ihrer eigenen „Silencing“-Maschinerie zugeführt wird und zur Ausschaltung der Expression eines essentiellen Insektengens führt (Zotti et al., 2018). In welchem Maß auch die bereits in der Pflanze aufgrund von Prozessierung entstandenen siRNAs vom Schadorganismus aufgenommen werden und wirksam sind, ist bisher noch nicht umfänglich geklärt (Christiaens et al., 2018). Seit 2015 darf gentechnisch veränderter Mais mit einer auf dem RNAi-Wirkmechanismus beruhenden Resistenz gegen den Maiswurzelbohrer (Diabrotica virgifera) unter dem Namen SmartStax PRO (Head et al., 2017) unter anderem in den USA angebaut werden; seit 2017 hat der pflanzeneingebaute Insektenschutz von MON 87411-Mais auch eine Zulassung der amerikanischen Umweltbehörde US-EPA als „plant incorporated protectant (PIP)“. In der EU wurde für diese gentechnisch veränderte Maislinie die Zulassung zum Import, zur Verarbeitung und für die Verwendung als Lebens- und Futtermittel beantragt und nach einer positiven wissenschaftlichen Bewertung durch die Europäische Behörde für Lebensmittelsicherheit (EFSA GMO Panel 2018) am 26.7.2019 genehmigt. Ein Anbau dieser Maispflanzen, die durch den Einsatz von Gentechnik erzeugt wurden, ist in Europa jedoch nicht erlaubt und in absehbarer Zeit auch nicht zu erwarten. Tabelle 1 gibt eine Übersicht über bisher für den Einsatz im Pflanzenschutz entwickelte und in einigen Ländern zugelassene gentechnisch veränderte RNAi-Pflanzen.
Tab. 1. Beispiele von RNAi-Pflanzen für Pflanzenschutz-Anwendungen
Schadorganimus | Zielgen | GV-Pflanze | Referenz | Zulassungsstatus USA/EU |
Papaya-Ringflecken-Virus (PRSV) | PRSV-Hüllprotein | Papaya 55–1 | USA: Anbau, Lebens-/Futtermittel | |
Scharka-Virus (PPV) | PPV-Hüllprotein | Prunus domestica C-5 | USA: Anbau, Lebens-/Futtermittel | |
Maiswurzelbohrer (Diabrotica virgifera) | DvSnf7 | Mais MON 87411 | USA: Anbau, Lebens-/Futtermittel |
dsRNA bzw. siRNA als RNAi-Auslöser können entweder innerhalb einer Zelle (endogen) gebildet werden, wie bei den oben erwähnten spontanen RNAi-Mutationen oder bei GV-Pflanzen, die aufgrund einer entsprechenden Genkonstruktion dsRNA produzieren, oder sie kann von außen (exogen) appliziert werden. Damit kann das RNAi-Wirkprinzip außer durch gentechnische Veränderung von Pflanzen auch auf anderem Weg zur Anwendung kommen. Die steigende Bedeutung von RNAi für den Pflanzenschutz durch externe dsRNA-Applikationen spiegelt sich in den Aktivitäten der europäischen COST-Action „iPLANTA“ (CA15223; https://iplanta.univpm.it/) wider (Mezzetti et al., 2020). Während dabei ursprünglich die Produktion von siRNAs durch entsprechende gentechnische Modifikation von Pflanzen im Vordergrund stand, wurden aufgrund der innerhalb der EU weiterhin fehlenden Akzeptanz von GV-Pflanzen zunehmend exogene RNA-Anwendungen thematisiert und ihr Entwicklungspotential analysiert (Taning et al., 2019). Ein weiteres Indiz für die internationale Relevanz von RNA-basierten extern applizierten Pflanzenschutzmitteln ist die Etablierung einer Ad hoc Expertengruppe zu RNAi-basierten Pflanzenschutzmitteln innerhalb der „Working Group on Pesticides“ der Organisation für wirtschaftliche Zusammenarbeit und Entwicklung OECD. Eine Zusammenfassung der Diskussionen während einer OECD-Konferenz in 2019 zu diesem Thema wurde kürzlich ebenso veröffentlicht (Mendelsohn et al., 2020) wie ein umfangreiches Dokument zur Umweltrisikobewertung der Anwendung von dsRNA-Pflanzenschutzmitteln (OECD, 2020).
Sequenzspezifische dsRNA für die Anwendung als biochemisches Pflanzenschutzmittel kann durch chemische Synthese, durch in vitro Transkription, in Bakterienzellkulturen oder in speziellen zellfreien Systemen hergestellt werden (Taning et al., 2019; Niehl et al., 2018). Während bei den recht teuren Verfahren der chemischen Synthese und der in vitro Transkription wie auch bei den meisten bakteriellen Produktionssystemen zunächst RNA-Einzelstränge synthetisiert werden, aus denen durch anschließende Hybridisierung komplementärer Stränge dsRNA entsteht, wurde von Niehl et al. (2018) ein RNA-Bakteriophagen-basierter in vivo-Ansatz zur direkten dsRNA-Synthese mittels Phagen-eigener RNA-abhängiger RNA-Polymerase entwickelt. Bei allen Herstellungsprozessen ist eine sorgfältige Qualitätskontrolle notwendig, um potentielle Kontaminationen zu verhindern und die Reinheit der dsRNA sicherzustellen. Bei der mikrobiellen Produktion ist darüber hinaus darauf zu achten, dass keine lebenden gentechnisch veränderten Mikroorganismen im Endprodukt vorhanden sind. Insbesondere durch die Entwicklung zellfreier Synthese-Plattformen ist mittlerweile eine kostengünstige Produktion ausreichender dsRNA-Mengen für Praxis-Anwendungen ohne den Einsatz gentechnisch veränderter Mikroorganismen möglich (Taning et al., 2019). Da dsRNA bei der Anwendung im Freiland zahlreichen zerstörerischen Umwelteinflüssen wie UV-Strahlung und mikrobiellen Nukleasen ausgesetzt ist, ist dabei in der Regel eine Stabilisierung durch entsprechende Formulierungen notwendig, um eine schnelle Inaktivierung zu verhindern (Bachmann et al., 2020; Taning et al., 2019). Eine weitere Einschränkung von dsRNA-Anwendungen im Pflanzenschutz ergibt sich daraus, dass sich Insekten sehr in ihrer Anfälligkeit gegenüber RNAi unterscheiden. Während bei Coleopteren generell eine gute Effizienz von RNAi-Applikationen zu beobachten ist, sind insbesondere Lepidopteren häufig insensitiv gegenüber oral zugeführter dsRNA (Zotti et al., 2018). Ursachen für unzureichende RNAi-Effizienz bei Insekten können dsRNA-abbauende Nukleasen im Speichel und Verdauungstrakt, fehlende zelluläre Aufnahmemechanismen, eine mangelhafte Silencing-Maschinerie sowie eine beeinträchtigte systemische Ausbreitung von siRNAs innerhalb des Insekts sein (Cooper et al., 2018).
Für die exogene Applikation von dsRNA zur spezifischen Kontrolle von Schädlingen, phytopathogenen Pilzen oder Viren stehen in Abhängigkeit vom Schadorganismus prinzipiell verschiedene Methoden zur Verfügung (Dalakouras et al., 2020). Im einfachsten Fall, wenn keine Aufnahme in das Pflanzeninnere notwendig ist, wie etwa bei der Bekämpfung von Blatt-fressenden Insekten, genügt eine Sprühapplikation (SIGS; spray-induced gene silencing) der Blätter mit dsRNA. Aufgrund mehrerer Barrieren, wie die Cuticula und die Pflanzenzellwand, bei intaktem, nicht verletztem Pflanzengewebe verbleiben RNA-Moleküle bei dieser Applikationsmethode größtenteils auf der Pflanzenoberfläche (Bachmann et al., 2020; Dalakouras et al., 2020). Die durch Sprays applizierte dsRNA wird von den Insekten oral aufgenommen, unabhängig von einer vorherigen Aufnahme in die Pflanze. Unter anderem sind dsRNA-Präparate zur Blattapplikation gegen Kartoffelkäfer (Leptinotarsa decemlineata) in der Testung und Entwicklung (San Miguel & Scott, 2015; Mehlhorn et al., 2020). Auch phytopathogene Pilze können durch SIGS kontrolliert werden, wie von Koch et al. (2016) für Fusarium graminearum in Gerste und von Wang et al. (2017) für Botrytis cinerea auf Ernteprodukten gezeigt wurde. Wirksame dsRNAs werden dabei von den Pilzzellen direkt aus der Umwelt aufgenommen; in bestimmten Fällen (z. B. bei Verwundung der Pflanze) wurde auch eine vorherige Aufnahme in die Pflanze und ein Transport in den Leitbahnen und damit eine systemische Wirkung in der Pflanze beobachtet (Koch et al., 2016).
Sollen Xylemsaft-saugende Insekten bekämpft werden, wie etwa Schaumzikaden, die in Europa als Hauptüberträger des Feuerbakteriums Xylella fastidiosa fungieren, sind aufwändigere Methoden notwendig, um die dsRNA an den Wirkungsort zu bringen. Möglich wäre eine Injektion ins Xylem der Pflanzen. Dalakouras et al. (2018) konnten in einem experimentellen Ansatz mit einem Markergen zeigen, dass doppelsträngige hairpin (hp) RNA durch Stammbohrung ins Xylem von holzigen Pflanzen (Malus domestica und Vitis vinifera) gelangte und dort systemisch transportiert wurde. Da der Transport apoplastisch und damit extrazellulär erfolgte, wurde die dsRNA in der Pflanze nicht in siRNAs prozessiert. Die fehlende Prozessierung in der Pflanze könnte vermutlich eine gute Bekämpfung der Insekten ermöglichen, da unter anderem von Bolognesi et al. (2012) gezeigt wurde, dass eine effiziente Silencing-Aktivität von der Aufnahme ausreichender Mengen unprozessierter dsRNA abhängig ist.
Für die Kontrolle von pflanzenpathogenen Viren muss die Effektor-RNA in die Pflanzenzellen gelangen und dort, falls dsRNA appliziert wird, prozessiert werden. Damit dsRNAs oder auch kleine siRNAs die Cuticula und die pflanzliche Zellwand effizient passieren können, muss entweder das Pflanzengewebe mechanisch verletzt werden, beispielsweise durch Hochdruck-Sprühapplikation (Dalakouras et al., 2016), oder die RNA muss an geeignete Trägersubstanzen gekoppelt werden (Jiang et al., 2014; Numata et al., 2014; Zhang et al., 2019). Eine erfolgreiche RNAi-basierte Bekämpfung von Pflanzenviren wurde unter anderem durch Spray-Applikation von Mais (Gan et al., 2010) oder mechanische Inokulation von Papaya (Shen et al., 2014) mit Rohextrakten aus dsRNA-produzierenden Bakterien erreicht. Neuere Ansätze mit dem Ziel der Virusbekämpfung sind in der Regel frei von Bakterienzell-Bestandteilen, um Kontaminationen mit ungewollten Nebenprodukten aus der Fermentation zu vermeiden. Häufig wird die gereinigte dsRNA in Experimenten zusammen mit dem Ziel-Virus mechanisch co-inokuliert, wodurch sie zeitgleich mit dem Virus in die Zellen gelangt und die gewünschte Resistenz vermittelt (Kaldis et al., 2018; Niehl et al., 2018).
Für die Praxistauglichkeit von RNAi-basierten Pflanzenschutzmitteln unter agronomischen Bedingungen ist es erforderlich, dass die applizierte dsRNA ausreichend lange auf der Pflanze stabil ist, um genügend wirksam zu sein und um häufige Anwendungswiederholungen zu vermeiden. Unter Freilandbedingungen, wo RNA durch UV-Licht und Mikroorganismen degradiert und durch Regen abgewaschen wird, wurde bereits 3 Tage nach Blatt-Applikation eine 95 %ige Abnahme der dsRNA-Menge gefunden (Bachmann et al., 2020). Von Mitter et al. (2017) wurde eine Strategie zur Verminderung der Degradation entwickelt, indem die dsRNA an mehrlagige Doppelhydroxyd (LDH)-Nanopartikel als Träger gekoppelt wurde. LDH-Nanopartikel („BioClay“) werden in der Umwelt langsam degradiert, wobei die dsRNA nach und nach freigegeben wird. In den Untersuchungen von Mitter et al. (2017) konnte durch „BioClay“-Sprays bei Tabak die Dauer des Schutzes gegen eine Virusinfektion von 5 Tagen auf 20 Tage nach Applikation von virusspezifischer dsRNA erhöht werden.
RNAi stellt ein neuartiges Wirkprinzip für den Pflanzenschutz in Deutschland dar; darauf basierende landwirtschaftliche Anwendungen bedürfen daher einer besonderen Bewertung. Für gentechnisch veränderte Pflanzen existiert in der Europäischen Union (EU) eine Gesetzgebung mit entsprechenden Richtlinien und Verordnungen, in denen sowohl die Umweltsicherheit der Pflanzen als auch die Sicherheit von daraus hergestellten Lebens- und Futtermitteln geregelt werden. Diese EU-Gesetzgebung gilt auch für gentechnisch veränderte RNAi-Pflanzen. Auf RNA-Interferenz basierende GV-Sojabohnen mit verbesserter Fettsäure-Zusammensetzung sowie der bereits erwähnte Wurzelbohrer-resistente Mais wurden bereits in der EU auf der Grundlage der Verordnung (EC) 1829/2003 für den Import und für die Verarbeitung als Lebens- und Futtermittel zugelassen (EFSA GMO Panel 2012, 2013, 2018).
Angesichts der zunehmenden Bedeutung der RNAi-Techniken gab die europäische Behörde für Lebensmittelsicherheit EFSA zur Unterstützung der Risikobewertung von RNAi-Pflanzen drei mittlerweile veröffentlichte Literaturstudien in Auftrag (Pačes et al., 2017; Christiaens et al., 2018; Dávalos et al., 2019). Auch wenn, wie unter anderem aus diesen Studien hervorgeht, für die Bewertung von RNAi-Pflanzen einige Besonderheiten bezüglich der erforderlichen Daten gelten, sind die in den Leitlinien-Dokumenten der EFSA dargelegten Aspekte der Sicherheitsbewertung von GV-Pflanzen grundsätzlich anwendbar (Arpaia et al., 2020; Papadopoulou et al., 2020). Da ausgehend von der zur dsRNA-Bildung führenden gentechnischen Veränderung keine neuen Proteine codiert werden, sind diesbezügliche Daten nicht relevant für die Sicherheitsbewertung von Nahrungs- und Futtermitteln. Mögliche unbeabsichtigte negative Auswirkungen auf den pflanzlichen Metabolismus aufgrund von off-target-Effekten von siRNAs in der Pflanze können ebenso wie intendierte Effekte durch Inhaltsstoffanalysen und gegebenenfalls Fütterungsstudien detektiert werden (Arpaia et al., 2020; Casacuberta et al., 2015). Darüber hinaus sind auf Grundlage der EU-Durchführungsverordnung 503/2013 bioinformatische Analysen durchzuführen, um potentielle off-target-Gene im Pflanzengenom zu identifizieren. Dazu wurden vom EFSA GMO-Gremium Kriterien festgelegt, basierend auf derzeitigen Kenntnissen über Zielgenspezifitäten natürlich vorkommender pflanzlicher miRNAs (EFSA GMO Panel 2017). Aufgrund der Bedeutung von RNAi-Mechanismen für die Genregulierung und die Virusabwehr bei Tieren und Pflanzen sind dsRNAs und ihre Prozessierungsprodukte natürliche ubiquitäre Bestandteile von Nahrungs- und Futtermitteln (Dávalos et al., 2019). Die Exposition nach oraler Aufnahme von dsRNA/siRNA ist bei Menschen und Nutztieren aufgrund von Degradation und diverser weiterer Barrieren im Verdauungstrakt dennoch vernachlässigbar gering (O´Neill et al., 2011; Petrick et al., 2013), so dass nach Verzehr bzw. Fütterung von RNAi-Pflanzen keine ausreichend hohen siRNA-Mengen für eine mögliche Beeinflussung der Aktivität menschlicher oder tierischer Gene zu erwarten sind (Arpaia et al., 2020; Papadopoulou et al., 2020). Im Fall des Anbaus sind insbesondere bei RNAi-Pflanzen zur Krankheits- oder Schädlingsbekämpfung mögliche Auswirkungen auf Nicht-Ziel-Organismen (Non-target organisms, NTO) zu betrachten, die sich aufgrund von möglichen Sequenzhomologien zwischen siRNAs und transkribierten Genen von NTOs ergeben könnten. Dabei können bioinformatische Analysen die Risikobewertung unterstützen (Arpaia et al., 2020; Papadopoulou et al., 2020). Eine zusätzliche bzw. alternative Methode zur Untersuchung von non-target-Effekten ist die Auswahl von phylogenetisch mit dem Schadorganismus verwandten Organismen, um das Aktivitätsspektrum einer dsRNA zu bestimmen (z. B. Bachmann et al., 2013). Neben sequenzspezifischen sind auch mögliche dsRNA-induzierte nicht-sequenzspezifische Effekte bei der Risikoanalyse zu berücksichtigen. dsRNA wird unabhängig vom RNAi-Wirkmechanismus unter anderem bei Pflanzen und Insekten im Rahmen der Virusabwehr als Pathogen-assoziiertes molekulares Muster (PAMP) erkannt und kann antivirale Immunreaktionen auslösen (Niehl et al., 2016; McMenamin et al., 2018). Ein wichtiger Punkt bei der Risikoanalyse ist außerdem die Frage der Exposition, die bei gentechnisch veränderten RNAi-Pflanzen unter anderem abhängig ist von der Höhe und dem Ort der dsRNA-Expression in der Pflanze sowie der Stabilität der dsRNA in verschiedenen Umweltkompartimenten (Schiemann et al., 2019; Arpaia et al., 2020). Bei der Betrachtung relevanter Risikoszenarien und beim Design spezifischer Bioassays in stufenweisen NTO-Risikobewertungsverfahren ist auch die unterschiedliche Sensitivität potentieller NTOs gegenüber von außen zugeführter dsRNA zu berücksichtigen.
Für externe Anwendungen von dsRNA-Pflanzenschutzmitteln gibt es im Gegensatz zu gentechnisch veränderten PIP-Pflanzen bisher weltweit noch keine Zulassungen (Mendelsohn et al., 2020). Aufgrund der Bedeutung der RNAi-Technologie und der anstehenden Produktentwicklungen fand bereits 2014 eine vom US-amerikanischen „Federal Insecticide, Fungicide and Rodenticide Act (FIFRA) Scientific Advisory Panel (SAP)“ organisierte Expertenkonferenz zur Bewertung von Umwelt- und Gesundheitsrisiken von RNAi-basierten Pflanzenschutzmitteln statt, in der neben der Anwendung als PIP auch externe dsRNA-Applikationen betrachtet wurden und deren Diskussionen und Empfehlungen veröffentlicht wurden (FIFRA, 2014). Um harmonisierte Rahmenbedingungen für die Regulierung von RNAi-basierten Pflanzenschutzmitteln zu schaffen, wurde außerdem von der „OECD Working Group on Pesticides (WGP)“ in 2015 die bereits erwähnte Ad hoc Expertengruppe initiiert. Von dieser OECD-Expertengruppe wurde im April 2019 eine internationale Konferenz mit 57 Teilnehmern aus Wissenschaft, Industrie und Regierungsbehörden veranstaltet mit dem Ziel, neueste wissenschaftliche Daten und Erkenntnisse hinsichtlich des Potentials und der Regulierung von dsRNA-Pflanzenschutzprodukten zusammenzutragen (Mendelsohn et al., 2020). Erörtert wurden sowohl Auswirkungen auf die Umwelt als auch auf die menschliche Gesundheit. Umfangreiche Überlegungen zur Umweltrisikoanalyse sind in dem vor kurzem publizierten OECD-Dokument dargelegt, in dem auch die Beiträge und Diskussionen der Konferenz berücksichtigt wurden (OECD, 2020). Grundsätzlich sind bei der Risikobewertung von dsRNA-Pflanzenschutzmitteln ähnliche Parameter wie bei RNAi-Pflanzen zu betrachten, mit dem Focus zunächst auf der Wahrscheinlichkeit der Exposition und auf der Frage der Empfindlichkeit von NTOs gegenüber extern applizierter dsRNA. Ähnlich wie bei konventionellen Pflanzenschutzmitteln ergeben sich aus der Art und der Methode der Applikation aber noch weitere Aspekte, die bei der Risikobewertung zu berücksichtigen sind. Dazu gehört der Einfluss von stabilisierenden Trägersubstanzen oder Formulierungen auf die Persistenz der dsRNA in der Umwelt und auf die Aufnahme durch Nicht-Zielorganismen, wodurch sowohl das Ausmaß der Exposition als auch die Sensitivität von NTOs gegenüber exogener dsRNA verändert werden kann (Taning et al., 2019; Mendelsohn et al., 2020; OECD, 2020). Auch wenn hinsichtlich Umweltpersistenz und Toxizität generell die üblichen Testmethoden für Pflanzenschutzmittel angewendet werden können, ist beim Design von Studien zur Risikoanalyse außerdem zu berücksichtigen, dass dsRNA-basierte Pflanzenschutzmittel aufgrund ihres Wirkungsprinzips im Gegensatz zu konventionellen Pflanzenschutzmitteln meist erst mit zeitlicher Verzögerung wirksam sind (Mendelsohn et al., 2020; OECD, 2020). Zu bedenken ist hinsichtlich einer nachhaltigen Nutzung auch, dass ebenso wie bei konventionellen chemischen Pflanzenschutzmitteln auch bei dsRNA-Produkten Resistenzen bei Schaderregern selektioniert werden können (Khajuria et al., 2018), und daher möglichst frühzeitig Strategien zum Resistenzmanagement entwickelt werden sollten.
Pflanzenschutzmittel mit dem für diesen Anwendungsbereich neuen Wirkungsmechanismus RNA-Interferenz können aufgrund ihrer großen Spezifität und Selektivität als vielversprechende Alternative zu konventionellen Pflanzenschutzwirkstoffen betrachtet werden. Voraussetzung für ihre praktische Anwendung in der Landwirtschaft ist aber neben der Entwicklung kostengünstiger und praxistauglicher dsRNA-basierter Produkte die Etablierung von geeigneten Regulierungsstandards sowie von angepassten Testmethoden für die Risikoanalyse. Trotz des aktuell großen Bedarfs an alternativen Pflanzenschutzansätzen, insbesondere bei der Kontrolle von Schadinsekten, ist zum jetzigen Zeitpunkt eine Prognose zu den Zukunftsaussichten von RNAi-basierten Pflanzenschutzmitteln in der EU und in Deutschland noch nicht möglich.
Die Autorin erklärt, dass keine Interessenskonflikte vorliegen.
Alyokhin, A., M. BAker, D. Mota-Sanchez, G. Dively, E. Grafius, 2008: Colorado potato beetle resistance to insecticides. American Potato Research 85, 395-413, DOI: 10.1007/s12230-008-9052-0.
Arpaia, S., O. Christiaens, K. Giddings, H. Jones, B. Mezzetti, F. Moronta-Barrios, J.N. Perry, J.B. Sweet, C.N.T. Taning, G. Smagghe, A. Dietz-Pfeilstetter, 2020: Biosafety of GM crop plants expressing dsRNA: Data requirements and EU regulatory considerations. Frontiers in Plant Science 11 (940), DOI: 10.3389/fpls.2020.00940.
Axtell, M.J., J.O. westholm, E.C. Lai, 2011: Vive la différence: biogenesis and evolution of microRNAs in plants and animals. Genome Biology 12 (221), DOI: 10.1186/gb-2011-12-4-221.
Bachmann, P, R. Bolognesi, W.J. Moar, G.M. Mueller, M.S. Paradise, P. Ramaseshadri, J. Tan, J.P. Uffman, J. Warren, B.E. Wiggins, S.L. Levine, 2013: Characterization of the spectrum of insecticidal activity of a double-stranded RNA with targeted activity against Western Corn Rootworm (Diabrotica virgifera virgifera LeConte). Transgenic Research 22, 1207-1222, DOI: 10.1007/s11248-013-9716-5.
Bachman, P.M., K.M. Huizinga, P.D. Jensen, G. Mueller, J. Tan, J.P. Uffman, S.L. Levine, 2016: Ecological risk assessment for DvSnf7 RNA: A plant-incorporated protectant with targeted activity against western corn rootworm. Regulatory Toxicology and Pharmacology 81, 77-88.
Bachmann, P., J. Fischer, Z. Song, E. Urbanczyk-Wochniak, G. Watson, 2020: Environmental fate and dissipation of applied dsRNA in soil, aquatic systems, and plants. Frontiers in Plant Science 11, DOI: 10.3389/fpls.2020.00021.
Baulcombe, D., 2004: RNA silencing in plants. Nature 431, 356-363, DOI: 10.1038/nature02874.
Blevins, T., R. Rajeswaran, P.V. Shivaprasad, D. Beknazariants, A. Si-Ammour, H.-S. Park, F. Vazquez, D. Robertson, F. Meins jr., T. Hohn, M.M. Pooggin, 2006: Four plant Dicers mediate viral smallRNA biogenesis and DNA virus induced silencing. Nucleic Acids Research 34 (21), 6233-6246, DOI: 10.1093/nar/gkl886.
Bolognesi, R., P. Ramaseshadri, J. Anderson, P. Bachman, W. Clinton, R. Flannagan, O. Ilagan, C. Lawrence, S. Levine, W. Moar, G. Mueller, J. Tan, J. Uffman, E. Wiggins, G. Heck, G. Segers, 2012: Characterizing the mechanism of action of double-stranded RNA against Western corn rootworm (Diabrotica virgifera virgifera LeConte). PLOS ONE 7 (10), e47534.
Casacuberta, J.M., Y. Devos, P. du Jardin, M. Ramon, H. Vaucheret, F. Nogué, 2015: Biotechnological uses of RNAi in plants: risk assessment considerations. Trends in Biotechnology 33 (3), 145-147, DOI: 10.1016/j.tibtech.2014.12.003.
Chakraborty, S., A.C. Newton, 2011: Climate change, plant diseases and food security: an overview. Plant Pathology 60, 2-14, DOI: 10.1111/j.1365-3059.2010.02411.x.
Christiaens, O., T. Dzhambazova, K. Kostov, S. Arpaia, M.R. Joga, I. Urru, J. Sweet, G. Smagghe, 2018: Literature review of baseline information on RNAi to support the environmental risk assessment of RNAi-based GM plants. EFSA supporting publication 15:1424, DOI: 10.2903/sp.efsa.2018.EN-1424.
Cooper, A.M.W., K. Silver, J. Zhang, Y. Park, K.Y. Zhu, 2018: Molecular mechanisms influencing efficiency of RNA interference in insects. Pest Management Science 75, 18-28, DOI: 10.1002/ps.5126.
Dalakouras, A., M. Wassenegger, J.N. McMillan, V. Cardoza, I. Maegele, E. Dadami, M. Runne, G. Krczal, M. Wassenegger, 2016: Induction of silencing in plants by high-pressure spraying of in vitro-synthesized small RNAs. Frontiers in Plant Science 7, 1327, DOI: 10.3389/fpls.2016.01327.
Dalakouras, A., W. Jarausch, G. Buchholz, A. Bassler, M. Braun, T. Manthey, G. Krczal, M. Wassenegger, 2018: Delivery of hairpin RNAs and small RNAs into woody and herbaceous plants by trunk injection and petiole absorption. Frontiers in Plant Science 9, 1253, DOI: 10.3389/fpls.2018.01253.
Dalakouras, A., M. Wassenegger, E. Dadami, I. Ganopoulos, M.L. Pappas, K. Papadopoulou, 2020: Genetically modified organism-free RNA interference: exogenous application of RNA molecules in plants. Plant Physiology 182, 38-50, DOI: 10.1104/pp.19.00570.
Dávalos, A., R. Henriques, M.J. Latasa, M. Laparra, M. Coca, 2019: Literature review of baseline information on non-coding RNA (ncRNA) to support the risk assessment of ncRNA-based genetically modified plants for food and feed. EFSA supporting publication 16:220. DOI: 10.2903/sp.efsa.2018.EN-1688.
EFSA Panel on Genetically Modified Organisms (GMO), 2012: Scientific opinion on application (EFSA-GMO-NL-2010-78) for the placing on the market of herbicide tolerant, increased oleic acid, genetically modified soybean MON 87705 for food and feed uses, import and processing under regulation (EC) No 1829/2003 from Monsanto. EFSA Journal 10, 2909, DOI: 10.2903/j.efsa.2012.2909.
EFSA Panel on Genetically Modified Organisms (GMO), 2013: Scientific opinion on application (EFSA-GMO-NL-2007-45) for the placing on the market of herbicide tolerant, high-oleic acid, genetically modified soybean 305423 for food and feed uses, import and processing under regulation (EC) No 1829/2003 from Pioneer. EFSA Journal 11, 3499, DOI: 10.2903/j.efsa.2013.3499.
EFSA GMO Panel, 2017: Annex II of the minutes of the 118th GMO Plenary meeting: Internal note on the strategy and technical aspects for small RNA plant off-target bioinformatics studies (https://www.efsa.europa.eu/sites/default/files/event/171025-m.pdf).
EFSA Panel on Genetically Modified Organisms (GMO), 2018: Scientific opinion on the assessment of genetically modified maize MON 87411 for food and feed uses, import and processing, under Regulation (EC) No 1829/2003 (application EFSA-GMO-NL-2015-124). EFSA Journal 16 (6), 5310, DOI: 10.2903/j.efsa.2018.5310.
FIFRA Scientific Advisory Panel Meeting, 2014: A Set of Scientific Issues Being Considererd by the Environmental Protection Agency regarding RNAi Technology: Problem Formulation for Human Health and Ecological Risk Assessment. United States Environmental Protection Agency, Washington, DC, p. 75.
Fire, A., S. Xu, M.K. Montgomery, S.A. Kostas, S.E. Driver, C.C. Mello, 1998: Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature 391, 806-811.
Gan, D., J. Zhang, H. Jiang, T. Jiang, S. Zhu, B. Cheng, 2010: Bacterially expressed dsRNA protects maize against SCMV infection. Plant Cell Reports 29, 1261-1268.
Garrett, K.A., S.P. Dendy, E.E. Frank, M.N. Rouse, S.E. Travers, 2006: Climate change effects on plant disease: genomes to ecosystems. Annual Review of Phytopathology 44, 489-509.
Gonsalves, D., A. Vegas, V. Prasartsee, R. Drew, J.Y. Suzuki, S. Tripathi, 2006: Developing papaya to control papaya ringspot virus by transgenic resistance, intergeneric hybridization, and tolerance breeding. Plant Breeding Reviews 26, 35-78.
Hamilton, A.J. & D.C. Baulcombe, 1999: A species of small antinsense RNA in post-transcriptional gene silencing in plants. Science 286, 950-952.
Head, G.P., M.W. Carroll, S.P. Evans, D.M. Rule, A.R. Willse, T.L. Clark, N.P. Storer, R.D. Flannagan, L.W. Samuel, L.J. Meinke, 2017: Evaluation of SmartStax and SmartStax PRO maize against western corn rootworn and northern corn rootworm: efficacy and resistance management. Pest Management Science 73, 1883-1899, DOI: 10.1002/ps.4554.
Ibrahim, A.B., F.J.L. Aragão, 2015: RNAi-mediated resistance to viruses in genetically engineered plants: In: Plant Gene Silencing. Methods in Molecular Biology, vol 1287.Mysore, K., M. Senthil-Kumar (Eds.). New York, NY. Humana Press, p. 81-92, DOI: 10.1007/978-1-4939-2453-0_5.
Jiang, L., L. Ding, B. He, J. Shen, Z. Xu, M. Yin, X. Zhang, 2014: Systemic gene silencing in plants triggered by fluorescent nanoparticle-delivered double-stranded RNA. Nanoscale 6, 9965-9969.
Kaldis, A., M. Berbati, O. Melita, C. Reppa, M. Holeva, P. Otten, A. Voloudakis, 2018: Exogenously applied dsRNA molecules deriving from the Zucchini yellow mosaic virus (ZYMV) genome move systemically and protect cucurbits against ZYMV. Molecular Plant Pathology 19, 883-895.
Khajuria, C., S. Ivashuta, E. Wiggins, L. Flagel, W. Moar, M. Pleau, K. Miller, Y. Zhang, P. Ramaseshadri, C. Jiang, R. Hodge, P. Jensen, M. Chen, A. Gowda, B. McNulty, C. Vazquez, R. Bolognesi, J. Haas, G. Head, T. Clark, 2018: Development and characterization of the first dsRNA-resistant insect population from western corn rootworm, Diabrotica virgifera virgifera LeConte. PLOS ONE 13 (5): e0197059.
Koch, A., D. Biedenkopf, A. Furch, L. Weber, O. Rossbach, E. Abdellatef, L. Linicus, J. Johannsmeier, L. Jelonek, A. Goesmann, V. Cardoza, J. McMillan, T. Mentzel, K.-H. Kogel, 2016: An RNAi-based control of Fusarium graminearum infections through spraying of long dsRNAs involves a plant passage and is controlled by the fungal silencing machinery. PLoS Pathogens 12 (10), DOI: 10.1371/journal.ppat.1005901.
Kusaba, M., K. Miyahara, S. Iida, H. Fukuoka, T. Takano, H. Sassa, M. Nishimura, T. Nishio, 2003: Low glutelin content 1: a dominant mutation that suppresses the glutelin multigene family via RNA silencing in rice. Plant Cell 15, 1455-1467, DOI: 10.1105/tpc.011452.
McMenamin, A.J., K.F. Daughenbaugh, F. Parekh, M.C. Pizzorno, M.L. Flenniken, 2018: Honey bee and bumble bee antiviral defense. Viruses 10 (395), DOI: 10.3390/v10080395.
Mehlhorn, S.G., S. Geibel, G. Bucher, R. Nauen, 2020: Profiling of RNAi sensitivity after foliar dsRNA exposure in different European populations of Colorado potato beetle reveals a robust response with minor variability. Pesticide Biochemistry and Physiology 166, 104569, DOI: 10.1016/j.pestbp.2020.104569.
Mendelsohn, M., A. Gathmann, D. Kardassi, S. Sachana, E. Hopwood, A. Dietz-Pfeilstetter, S. Michelsen-Correa, S. Fletcher, A. Székács, 2020: Summary of discussions from 2019 OECD conference on RNAi based pesticides. Frontiers in Plant Science 11 (740). DOI: 10.3389/fpls.2020.00740.
Mezzetti, B., G. Smagghe, S. Arpaia, O. Christiaens, A. Dietz-Pfeilstetter, H. Jones, K. Kostov, S. Sabbadini, H.-G. Opsahl-Sorteberg, V. Ventura, C.N.T. Taning, J. Sweet, 2020: RNAi: What is ist position in agriculture? Journal of Pest Science 93 (4), 1125-1130. DOI: 10.1007/s10340-020-01238-2.
Mitter, N., E.A. Worrall, K.E. Robinson, P. Li, R.G. JaiN, C. Taochy, S.J. Fletcher, B.J Carroll, G.Q. Lu, Z.P. Xu, 2017: Clay nanosheets for topical delivery of RNAi for sustained protection against plant viruses. Nature Plants 3, DOI: 10.1038/nplants.2016.207.
Niehl, A., M. Soininen, M.M. Poranen, M. Heinlein, 2018: Synthetic biology approach for plant protection using dsRNA. Plant Biotechnology Journal 16, 1679-1687.
Niehl, A., I. Wyrsch, T. Boller, M. Heinlein, 2016: Double-stranded RNAs induce a pattern-triggered immune signalling pathway in plants. New Phytologist 211, 1008-1019.
Numata, K., M. Ohtani, T. Yoshizumi, T. Demura, Y. Kodama, 2014: Local gene silencing in plants via synthetic dsRNA and carrier peptide. Plant Biotechnology Journal 12, 1027-1034.
OECD, 2020: Considerations for the Environmental Risk Assessment of the Application of Sprayed or Externally Applied dsRNA-based Pesticides. Series on Pesticides, No. 104. Access: 5 October 2020, ENV/JM/MONO(2020)26.
O´Neill, M.J., L. Bourre, S. Melgar, C.M. O´Driscoll, 2011: Instestinal delivery of non-viral gene therapeutics: physiological barriers and preclinical models. Drug Discorvery Today 16, 203-218, DOI: 10.1016/j.drudis.2011.01.003.
Pačes, J., M. Nič, T. Novotný, P. Svoboda, 2017: Literature review of baseline information to support the risk assessment of RNAi-based GM plants. EFSA supporting publication 14: 1246, DOI: 10.2903/sp.efsa.2017.EN-1246.
Papadopoulou, N., Y. Devos, F. Álvarez-Alfageme, A. Lanzoni, E. Waigmann, 2020: Risk assessment considerations for genetically modified RNAi plants: EFSA´s activities and perspective. Frontiers in Plant Science 11 (445), DOI: 10.3389/fpls.2020.00445.
Petrick, J.S., B. Brower-Toland, A.L. Jackson, L.D. Kier, 2013: Safety assessment of food and feed from biotechnology-derived crops employing RNA-mediated gene regulation to achieve desired traits: A scientific review. Regulatory Toxicology and Pharmacology 66 (2), 157-176, DOI: 10.1016/j.yrtph.2013.03.008.
Redenbaugh, K., T. Berner, D. Emlay, B. Frankos, W. Hiatt, C. Houck, M. Kramer, L. Malyj, B. Martineau, N. Rachman, L. Rudenko, R. Sanders, R. Sheehy, R. Wixtrom, 1993: Regulatory issues for commercialization of tomatoes with an antisense polygalacturonase gene. In Vitro Cellular & Developmental Biology 29P, 17-26.
Rupp, S., R.W.S. Weber, D. Rieger, P. Detzel, M. Hahn, 2017: Spread of Botrytis cynerea strains with multiple fungicide resistance in german horticulture. Frontiers in Microbiology 7, 1-12.
San Miguel, K. & Scott, 2015: The next generation of insecticides: dsRNA is stable as a foliar-applied insecticide. Pest Management Science 72, 801-809.
Schiemann, J., A. Dietz-Pfeilstetter, F. Hartung, C. Kohl, J. Romeis, T. Sprink, 2019: Risk assessment and regulation of plants modified by modern biotechniques: Current status and future challenges. Annual Review of Plant Biology 70, 699-726, DOI: 10.1146/annurev-arplant-050718-100025.
Scorza, R., A. Callahan, C. Dardick, M. Ravelonandro, J. Polak, T. Malinowski, I. Zagrai, M. Cambra, I. Kamenova, 2013: Genetic engineering of Plum pos virus resistance: `HoneySwee´plum – from concept to product. Plant Cell Tissue and Organ Culture 115, 1-12, DOI: 10.1007/s11240-013-0339-6.
Shen, W., G. Yang, Y. Chen, P. Yan, D. Tuo, X. Li, P. Zhou, 2014: Resistance of non-transgenic papaya plants to papaya ringspot virus (PRSV) mediated by intron-containing hairpin dsRNAs expressed in bacteria. Acta Virologica 58, 261-266.
Taning, C.N.T., S. Arpaia, O. Christiaens, A. Dietz-Pfeilstetter, H. Jones, B. Mezzetti, S. Sabbadini, H.-G. Sorteberg, J. Sweet, V. Ventura, G. Smagghe, 2019: RNA-based biocontrol compounds: current status and perspectives to reach the market. Pest Management Science 76 (3), 841-845, DOI: 10.1002/ps.5686.
Tuteja, J.H., S.J. Clough, W.C. Chan, L.O. Vodkin, 2004: Tissue-specific gene silencing mediated by a naturally occurring chalcon synthase gene cluster in Glycine max. Plant Cell 16, 819-835.
Vaucheret, H., 2006: Post-transcriptional small RNA pathways in plants: mechanisms and regulations. Genes & Development 20, 759-771.
Wang, M., T. Nicholas, H. Jin, 2017: Cross-kingdom RNA trafficking and environmental RNAi for powerful innovative pre- and post-harvest plant protection. Current Opinion in Plant Biology 38, 133-141, DOI: 10.1016/j.pbi.2017.05.003.
Waltz, E., 2015: Nonbrowning GM apple cleared for market. Nature Biotechnology 33 (4), 326-327. DOI: 10.1038/nbt0415-326c.
Wernecke, A., M. Frommberger, R. Forster, J. Pistorius, 2019: Lethal effects of various tank mixtures including insecticides, fungicides and fertilizers on honey bees under laboratory, semi-field and field conditions. Journal of Consumer Protection and Food Safety 14 (3), 239-249, DOI: 10/1007/s00003-019-01233-5.
Zhang, H., G.S. Demirer, H. Zhang, T. Ye, N.S. Goh, A.J. Aditham, F.J. Cunningham, C. Fan, M.P. Landry, 2019: DNA nanostructures coordinate gene silencing in mature plants. Proceedings of the National Academy of Sciences of the United States of America 116 (15), 7543-7548, DOI: 10.1073/pnas.1818290116.
Zotti, M., E. Avila dos Santos, D. Cagliari, O. Christiaens, C.N.T. Taning, G. Smagghe, 2018: RNA interference technology in crop protection against arthropod pests, pathogens and nematodes. Pest Management Science 74 (6), 1239-1250, DOI: 10.1002/ps.4813.